Responsables : Anne Bigot et Vincent Mouly
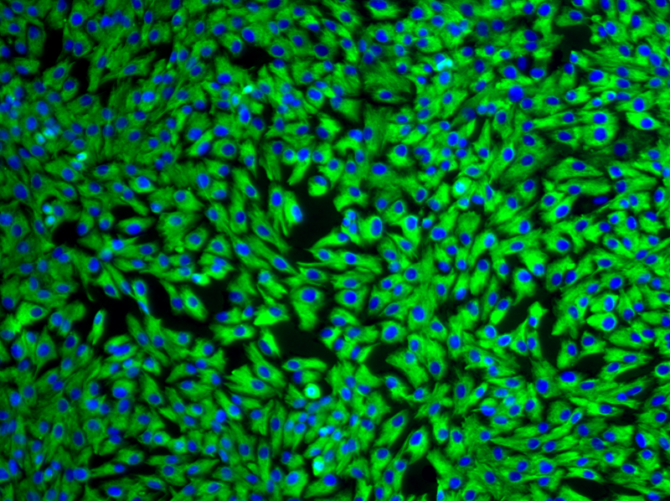
Avec le développement de nombreuses approches thérapeutiques innovantes pour les maladies neuromusculaires, ciblant l’ADN, l’ARN ou les protéines, les cellules musculaires isolées de patients, appelées myoblastes, représentent un modèle in vitro idéal pour évaluer ces approches : elles fournissent un matériel facile à cribler ou à évaluer l’efficacité tout en réduisant les expérimentations animales. Il est important de noter qu’elles portent également la mutation exacte du patient dans son propre environnement génétique. Ces approches in vitro ont des limites : les cellules somatiques humaines ont une capacité de prolifération limitée, régulée par l’horloge mitotique, et atteignent la sénescence réplicative après un nombre défini de divisions. Cette limite de prolifération est atteinte encore plus tôt dans les maladies dégénératives. Nous avons neutralisé la sénescence réplicative des cellules musculaires humaines en utilisant une double transduction avec des vecteurs lentiviraux, créant ainsi des lignées de cellules humaines immortelles.
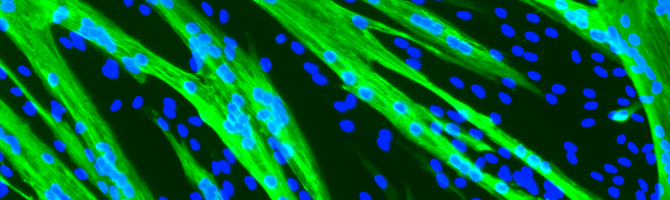
 La plateforme d’immortalisation de cellules humaines de l’Institut de Myologie a généré plus de 180 lignées de myoblastes humains isolés de patients souffrant de plus de 36 maladies différentes (DMD, LGMD, FSHD, SMA…). L’accès aux biopsies musculaires pouvant être limité pour certaines pathologies, nous avons également développé l’immortalisation de fibroblastes cutanés. Ces fibroblastes immortalisés sont ensuite transduits par un facteur de transcription inductible MyoD, un facteur de transcription spécifique du muscle, et ces cellules myoconverties forment des myotubes et expriment des marqueurs musculaires comme les cellules musculaires.
La plateforme d’immortalisation de cellules humaines de l’Institut de Myologie a généré plus de 180 lignées de myoblastes humains isolés de patients souffrant de plus de 36 maladies différentes (DMD, LGMD, FSHD, SMA…). L’accès aux biopsies musculaires pouvant être limité pour certaines pathologies, nous avons également développé l’immortalisation de fibroblastes cutanés. Ces fibroblastes immortalisés sont ensuite transduits par un facteur de transcription inductible MyoD, un facteur de transcription spécifique du muscle, et ces cellules myoconverties forment des myotubes et expriment des marqueurs musculaires comme les cellules musculaires.
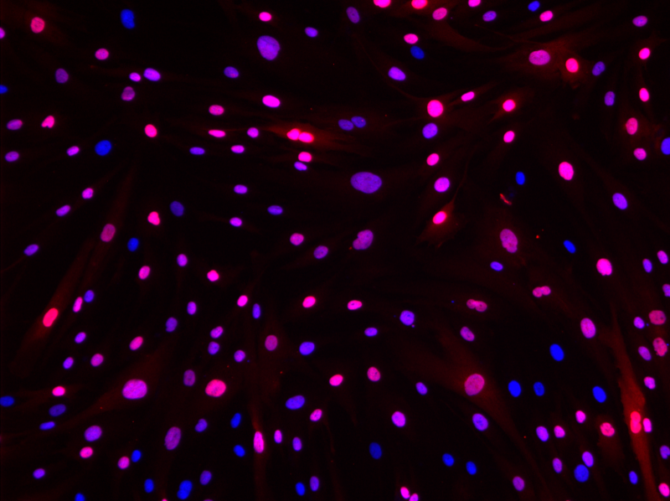
 La perte de masse musculaire chez les patients atteints de maladies neuromusculaires s’accompagne souvent de son remplacement par du tissu fibreux et/ou adipeux. Ces dernières années, la recherche dans ce domaine s’est concentrée sur le rôle joué par les progéniteurs fibro-adipogéniques (FAPs) en raison de leur capacité à se différencier en adipocytes ou en cellules fibroblastiques. Pour soutenir cette recherche, MyoLine développe actuellement des modèles de FAPs isolés à partir de différentes dystrophies musculaires.
La perte de masse musculaire chez les patients atteints de maladies neuromusculaires s’accompagne souvent de son remplacement par du tissu fibreux et/ou adipeux. Ces dernières années, la recherche dans ce domaine s’est concentrée sur le rôle joué par les progéniteurs fibro-adipogéniques (FAPs) en raison de leur capacité à se différencier en adipocytes ou en cellules fibroblastiques. Pour soutenir cette recherche, MyoLine développe actuellement des modèles de FAPs isolés à partir de différentes dystrophies musculaires.
Depuis plusieurs années, ces lignées cellulaires sont mises à la disposition de la communauté scientifique sur une base collaborative, et de nombreux laboratoires internationaux les utilisent déjà. Lorsque le consentement signé par le donneur le permet, les lignées cellulaires peuvent être également utilisées dans le cadre d’accords de transfert de matériel pour le développement d’outils thérapeutiques par des partenaires privés.
Exemples de lignées cellulaires
Envoyez vos demandes à anne.bigot@sorbonne-universite.fr & vincent.mouly@sorbonne-universite

Composition de l’équipe
Bensalah Mona, Ingénieure de recherche
Bigot Anne, Chargée de recherche
Diarra Rokiatou, Apprentie
Mamchaoui Kamel, Ingénieur de recherche
Mouly Vincent, Directeur de recherche
Ohana Jessica, Ingénieure d’études
Nos dernières publications
- HSPB8 frameshift mutant aggregates weaken chaperone-assisted selective autophagy in neuromyopathies. Tedesco B, Vendredy L, Adriaenssens E, Cozzi M, Asselbergh B, Crippa V, Cristofani R, Rusmini P, Ferrari V, Casarotto E, Chierichetti M, Mina F, Pramaggiore P, Galbiati M, Piccolella M, Baets J, Baeke F, De Rycke R, Mouly V, Laurenzi T, Eberini I, Vihola A, Udd B, Weiss L, Kimonis V, Timmerman V, Poletti A. Autophagy. 2023 Aug;19(8):2217-2239. doi: 10.1080/15548627.2023.2179780. Epub 2023 Feb 28. PMID: 36854646 Free PMC article.
- Caveolae and Bin1 form ring-shaped platforms for T-tubule initiation. Lemerle E, Lainé J, Benoist M, Moulay G, Bigot A, Labasse C, Madelaine A, Canette A, Aubin P, Vallat JM, Romero NB, Bitoun M, Mouly V, Marty I, Cadot B, Picas L, Vassilopoulos S. Elife. 2023 Apr 21;12:e84139. doi: 10.7554/eLife.84139. PMID: 37083699 Free PMC article.
- Prime editing strategies to mediate exon skipping in DMD gene. Happi Mbakam C, Roustant J, Rousseau J, Yameogo P, Lu Y, Bigot A, Mamchaoui K, Mouly V, Lamothe G, Tremblay JP. Front Med (Lausanne). 2023 May 25;10:1128557. doi: 10.3389/fmed.2023.1128557. eCollection 2023. PMID: 37305116 Free PMC article.
- Aberrant Adenosine Triphosphate Release and Impairment of P2Y2-Mediated Signaling in Sarcoglycanopathies. Benzi A, Baratto S, Astigiano C, Sturla L, Panicucci C, Mamchaoui K, Raffaghello L, Bruzzone S, Gazzerro E, Bruno C. Lab Invest. 2023 Mar;103(3):100037. doi: 10.1016/j.labinv.2022.100037. Epub 2023 Jan 10. PMID: 36925196
- High-capacity adenovector delivery of forced CRISPR-Cas9 heterodimers fosters precise chromosomal deletions in human cells. Tasca F, Brescia M, Liu J, Janssen JM, Mamchaoui K, Gonçalves MAFV. Mol Ther Nucleic Acids. 2023 Feb 22;31:746-762. doi: 10.1016/j.omtn.2023.02.025. eCollection 2023 Mar 14. PMID: 36937620
- Tetraspanin CD82 Associates with Trafficking Vesicle in Muscle Cells and Binds to Dysferlin and Myoferlin. Fontelonga T, Hall AJ, Brown JL, Jung YL, Alexander MS, Dominov JA, Mouly V, Vieira N, Zatz M, Vainzof M, Gussoni E. Adv Biol (Weinh). 2023 Jul 12:e2300157. doi: 10.1002/adbi.202300157. Online ahead of print. PMID: 37434585
- Bioengineering a miniaturized in vitro 3D myotube contraction monitoring chip to model muscular dystrophies. Rose N, Estrada Chavez B, Sonam S, Nguyen T, Grenci G, Bigot A, Muchir A, Ladoux B, Cadot B, Le Grand F, Trichet L. Biomaterials. 2023 Feb;293:121935. doi: 10.1016/j.biomaterials.2022.121935. Epub 2022 Dec 13. PMID: 36584444
- Cellular and Genomic Features of Muscle Differentiation from Isogenic Fibroblasts and Myoblasts. Benarroch L, Madsen-Østerbye J, Abdelhalim M, Mamchaoui K, Ohana J, Bigot A, Mouly V, Bonne G, Bertrand AT, Collas P. Cells. 2023 Aug 3;12(15):1995. doi: 10.3390/cells12151995. PMID: 37566074 Free PMC article.
- Muscle Specific Promotors for Gene Therapy – A Comparative Study in Proliferating and Differentiated Cells. Dietz J, Jacobsen F, Zhuge H, Daya N, Bigot A, Zhang W, Ehrhardt A, Vorgerd M, Ehrke-Schulz E. J Neuromuscul Dis. 2023;10(4):575-592. doi: 10.3233/JND-221574. PMID: 37270809 Free PMC article.
- Targeting Duchenne muscular dystrophy by skipping DMD exon 45 with base editors. Gapinske M, Winter J, Swami D, Gapinske L, Woods WS, Shirguppe S, Miskalis A, Busza A, Joulani D, Kao CJ, Kostan K, Bigot A, Bashir R, Perez-Pinera P. Mol Ther Nucleic Acids. 2023 Jul 27;33:572-586. doi: 10.1016/j.omtn.2023.07.029. eCollection 2023 Sep 12. PMID: 37637209 Free PMC article.
- Autosomal dominant in cis D4Z4 repeat array duplication alleles in facioscapulohumeral dystrophy. Lemmers RJLF, Butterfield R, van der Vliet PJ, de Bleecker JL, van der Pol L, Dunn DM, Erasmus CE, D’Hooghe M, Verhoeven K, Balog J, Bigot A, van Engelen B, Statland J, Bugiardini E, van der Stoep N, Evangelista T, Marini-Bettolo C, van den Bergh P, Tawil R, Voermans NC, Vissing J, Weiss RB, van der Maarel SM. Brain. 2023 Sep 13:awad312. doi: 10.1093/brain/awad312. Online ahead of print. PMID: 37703328
- Identification of a muscle-specific isoform of VMA21 as a potent actor in X-linked myopathy with excessive autophagy pathogenesis. Cocchiararo I, Cattaneo O, Rajendran J, Chabry F, Cornut M, Soldati H, Bigot A, Mamchaoui K, Gibertini S, Bouche A, Ham DJ, Laumonier T, Prola A, Castets P. Hum Mol Genet. 2023 Sep 26:ddad164. doi: 10.1093/hmg/ddad164. Online ahead of print. PMID: 37756622
- CRISPR-Cas9 KO Cell Line Generation and Development of a Cell-Based Potency Assay for rAAV-FKRP Gene Therapy. Geoffroy M, Pili L, Buffa V, Caroff M, Bigot A, Gicquel E, Rouby G, Richard I, Fragnoud R. Cells. 2023 Oct 12;12(20):2444. doi: 10.3390/cells12202444. PMID: 37887288 Free PMC article.
- CRISPR-Cas9 editing of a TNPO3 mutation in a muscle cell model of limb-girdle muscular dystrophy type D2. Poyatos-García J, Blázquez-Bernal Á, Selva-Giménez M, Bargiela A, Espinosa-Espinosa J, Vázquez-Manrique RP, Bigot A, Artero R, Vilchez JJ. Mol Ther Nucleic Acids. 2023 Jan 11;31:324-338. doi: 10.1016/j.omtn.2023.01.004. eCollection 2023 Mar 14. PMID: 36789274 Free PMC article.
- A homozygous loss of function variant in POPDC3: From invalidating exercise intolerance to a limb-girdle muscular dystrophy phenotype. De Ridder W, de Vries G, Van Schil K, Deconinck T, Mouly V, Straub V, Baets J. Neuromuscul Disord. 2023 May;33(5):432-439. doi: 10.1016/j.nmd.2023.04.003. Epub 2023 Apr.