Responsable : Anne Bigot
Avec le développement de nombreuses approches thérapeutiques innovantes pour les maladies génétiques, ciblant l’ADN, l’ARN ou les protéines, les cellules musculaires isolées de patients, appelées myoblastes, représentent un modèle in vitro idéal pour évaluer ces approches sur les maladies neuromusculaires. Ces cellules ont l’avantage d’être simples d’utilisation, de diminuer le nombre d’expérimentation sur les animaux mais également de porter la mutation exacte du patient dans son propre environnement génétique. Ces approches in vitro ont cependant des limites : les cellules somatiques humaines ont une capacité de prolifération limitée, régulée par l’horloge mitotique, et atteignent la sénescence réplicative après un nombre défini de divisions. Cette limite de prolifération est atteinte encore plus tôt dans les maladies dégénératives. MyoLine neutralise la sénescence réplicative des cellules musculaires humaines en utilisant une double transduction avec des vecteurs lentiviraux, créant ainsi des lignées de cellules humaines immortelles.
La plateforme d’immortalisation de cellules humaines de l’Institut de Myologie a généré ainsi depuis sa création en 2007 plus de 200 lignées de myoblastes humains isolés de patients souffrant de plus de 36 maladies différentes (DMD, LGMD, FSHD, SMA…). L’accès aux biopsies musculaires pouvant être limité pour certaines pathologies, nous avons également développé l’immortalisation de fibroblastes cutanés. Ces fibroblastes immortalisés sont ensuite transduits par un facteur de transcription inductible MyoD, et ces cellules myoconverties forment des myotubes et expriment des marqueurs musculaires comme les cellules musculaires.
La perte de masse musculaire chez les patients atteints de maladies neuromusculaires s’accompagne souvent de son remplacement par du tissu fibreux et/ou adipeux. Ces dernières années, la recherche dans ce domaine s’est concentrée sur le rôle joué par les progéniteurs fibro-adipogéniques (FAPs) en raison de leur capacité à se différencier en adipocytes ou en cellules fibroblastiques. Pour soutenir cette recherche, MyoLine développe actuellement des modèles de FAPs isolés à partir de différentes dystrophies musculaires.
Depuis plusieurs années, ces lignées cellulaires sont mises à la disposition de la communauté scientifique sur une base collaborative, et de nombreux laboratoires internationaux les utilisent déjà. Lorsque le consentement signé par le donneur le permet, les lignées cellulaires peuvent être également utilisées dans le cadre d’accords de transfert de matériel pour le développement d’outils thérapeutiques par des partenaires privés.
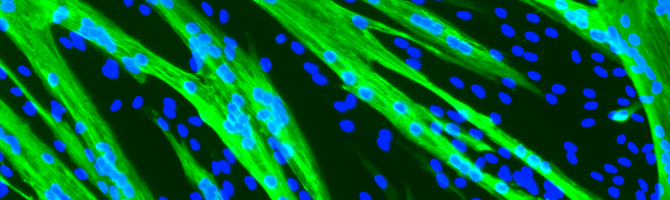
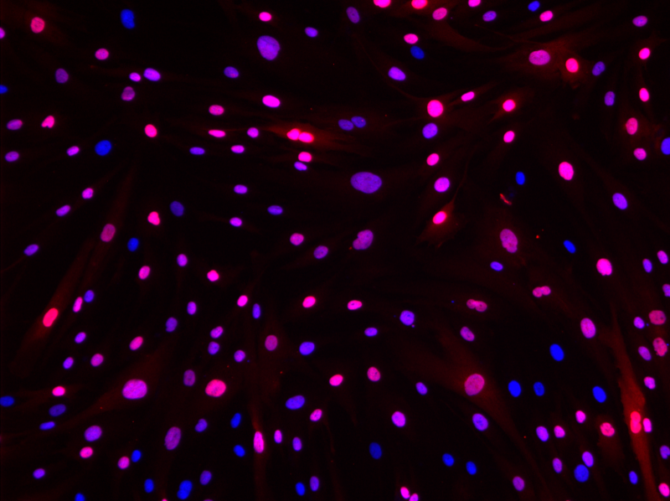
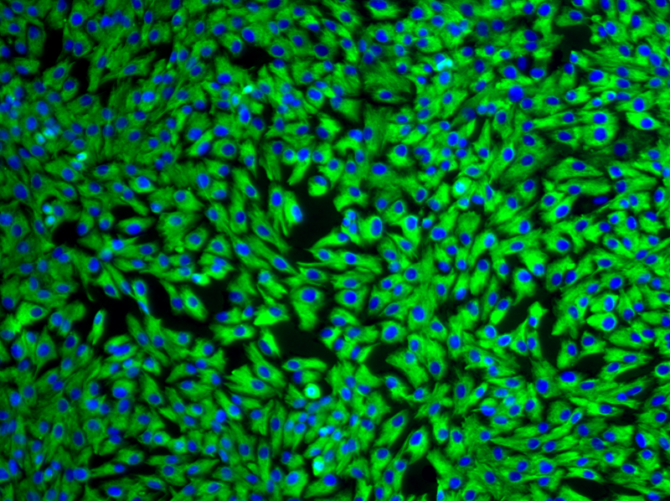
Exemples de lignées cellulaires
Envoyez vos demandes à anne.bigot@sorbonne-universite.fr & vincent.mouly@sorbonne-universite.fr

Composition de l’équipe
Bigot Anne, Cheffe de projet de Recherche, Responsable Plateforme
Bensalah Mona, Ingénieure de Recherche
Diarra Rokiatou, Apprentie
Filachet Alexis, Apprenti
Mamchaoui Kamel, Ingénieur de Recherche
Mouly Vincent, Directeur de Recherche Emérite
Ohana Jessica, Ingénieure d’études
Publications 2024
- Bin Haidar H, Almeida JR, Williams J, Guo B, Bigot A, Senthilkumaran S, Vaiyapuri S, Patel K. Differential effects of the venoms of Russell’s viper and Indian cobra on human myoblasts. Sci Rep. 2024 Feb 7;14(1):3184. doi: 10.1038/s41598-024-53366-9. PMID: 38326450 Free PMC article.
- Lemmers RJLF, Butterfield R, van der Vliet PJ, de Bleecker JL, van der Pol L, Dunn DM, Erasmus CE, D’Hooghe M, Verhoeven K, Balog J, Bigot A, van Engelen B, Statland J, Bugiardini E, van der Stoep N, Evangelista T, Marini-Bettolo C, van den Bergh P, Tawil R, Voermans NC, Vissing J, Weiss RB, van der Maarel SM. Autosomal dominant in cis D4Z4 repeat array duplication alleles in facioscapulohumeral dystrophy. Brain. 2024 Feb 1;147(2):414-426. doi: 10.1093/brain/awad312. PMID: 37703328 Free PMC article.
- De Spiegeleer A, Descamps A, Wynendaele E, Naumovski P, Crombez L, Planas M, Feliu L, Knappe D, Mouly V, Bigot A, Bielza R, Hoffmann R, Van Den Noortgate N, Elewaut D, De Spiegeleer B. Streptococcal quorum sensing peptide CSP-7 contributes to muscle inflammation and wasting. Biochim Biophys Acta Mol Basis Dis. 2024 Apr;1870(4):167094. doi: 10.1016/j.bbadis.2024.167094. Epub 2024 Feb 29. PMID: 38428683
- Galli F, Bragg L, Rossi M, Proietti D, Perani L, Bacigaluppi M, Tonlorenzi R, Sibanda T, Caffarini M, Talapatra A, Santoleri S, Meregalli M, Bano-Otalora B, Bigot A, Bozzoni I, Bonini C, Mouly V, Torrente Y, Cossu G. Cell-mediated exon skipping normalizes dystrophin expression and muscle function in a new mouse model of Duchenne Muscular Dystrophy. EMBO Mol Med. 2024 Apr;16(4):927-944. doi: 10.1038/s44321-024-00031-3. Epub 2024 Mar 4. PMID: 38438561 Free PMC article.
- Yin A, Fu W, Elengickal A, Kim J, Liu Y, Bigot A, Mamchaoui K, Call JA, Yin H. Chronic hypoxia impairs skeletal muscle repair via HIF-2α stabilization. J Cachexia Sarcopenia Muscle. 2024 Apr;15(2):631-645. doi: 10.1002/jcsm.13436. Epub 2024 Feb 9. PMID: 38333911 Free PMC article.
- Handal T, Juster S, Abu Diab M, Yanovsky-Dagan S, Zahdeh F, Aviel U, Sarel-Gallily R, Michael S, Bnaya E, Sebban S, Buganim Y, Drier Y, Mouly V, Kubicek S, van den Broek WJAA, Wansink DG, Epsztejn-Litman S, Eiges R. Differentiation shifts from a reversible to an irreversible heterochromatin state at the DM1 locus. Nat Commun. 2024 Apr 16;15(1):3270. doi: 10.1038/s41467-024-47217-4. PMID: 38627364 Free PMC article.
- Granados A, Zamperoni M, Rapone R, Moulin M, Boyarchuk E, Bouyioukos C, Del Maestro L, Joliot V, Negroni E, Mohamed M, Piquet S, Bigot A, Le Grand F, Albini S, Ait-Si-Ali S. SETDB1 modulates the TGFβ response in Duchenne muscular dystrophy myotubes. Sci Adv. 2024 May 3;10(18):eadj8042. doi: 10.1126/sciadv.adj8042. Epub 2024 May 1. PMID: 38691608 Free PMC article.
- Núñez-Manchón J, Capó J, Martínez-Piñeiro A, Juanola E, Pesovic J, Mosqueira-Martín L, González-Imaz K, Maestre-Mora P, Odria R, Savic-Pavicevic D, Vallejo-Illarramendi A, Mamchaoui K, Bigot A, Mouly V, Suelves M, Nogales-Gadea G. Immortalized human myotonic dystrophy type 1 muscle cell lines to address patient heterogeneity. iScience. 2024 May 7;27(6):109930. doi: 10.1016/j.isci.2024.109930. eCollection 2024 Jun 21. PMID: 38832025 Free PMC article.
- Engal E, Sharma A, Aviel U, Taqatqa N, Juster S, Jaffe-Herman S, Bentata M, Geminder O, Gershon A, Lewis R, Kay G, Hecht M, Epsztejn-Litman S, Gotkine M, Mouly V, Eiges R, Salton M, Drier Y. DNMT3B splicing dysregulation mediated by SMCHD1 loss contributes to DUX4 overexpression and FSHD pathogenesis. Sci Adv. 2024 May 31;10(22):eadn7732. doi: 10.1126/sciadv.adn7732. Epub 2024 May 29. PMID: 38809976 Free PMC article.
- Moreno N, Sabater-Arcis M, Sevilla T, Alonso MP, Ohana J, Bargiela A, Artero R. Therapeutic potential of oleic acid supplementation in myotonic dystrophy muscle cell models. Biol Res. 2024 May 17;57(1):29. doi: 10.1186/s40659-024-00496-z. PMID: 38760841 Free PMC article.
- Ghosh S, Arshi MU, Ghosh S, Jash M, Sen S, Mamchaoui K, Bhattacharyya S, Rana NK, Ghosh S. –Discovery of Quinazoline and Quinoline-Based Small Molecules as Utrophin Upregulators via AhR Antagonism for the Treatment of Duchenne Muscular Dystrophy. J Med Chem. 2024 Jun 13;67(11):9260-9276. doi: 10.1021/acs.jmedchem.4c00398. Epub 2024 May 21. PMID: 38771158
- Beaufils M, Melka M, Brocard J, Benoit C, Debbah N, Mamchaoui K, Romero NB, Dalmas-Laurent AF, Quijano-Roy S, Fauré J, Rendu J, Marty I. Functional benefit of CRISPR-Cas9-induced allele deletion for RYR1 dominant mutation. Mol Ther Nucleic Acids. 2024 Jun 17;35(3):102259. doi: 10.1016/j.omtn.2024.102259. eCollection 2024 Sep 10. PMID: 39071953 Free PMC article.
- Rich J, Bennaroch M, Notel L, Patalakh P, Alberola J, Issa F, Opolon P, Bawa O, Rondof W, Marchais A, Dessen P, Meurice G, Le-Gall M, Polrot M, Ser-Le Roux K, Mamchaoui K, Droin N, Raslova H, Maire P, Geoerger B, Pirozhkova I. DiPRO1 distinctly reprograms muscle and mesenchymal cancer cells. EMBO Mol Med. 2024 Aug;16(8):1840-1885. doi: 10.1038/s44321-024-00097-z. Epub 2024 Jul 15. PMID: 39009887 Free PMC article.
- Grandi FC, Astord S, Pezet S, Gidaja E, Mazzucchi S, Chapart M, Vasseur S, Mamchaoui K, Smeriglio P. Characterization of SMA type II skeletal muscle from treated patients shows OXPHOS deficiency and denervation. JCI Insight. 2024 Sep 12;9(20):e180992. doi: 10.1172/jci.insight.180992. PMID: 39264856 Free PMC article.
- Lemoine J, Dubois A, Dorval A, Jaber A, Warthi G, Mamchaoui K, Wang T, Corre G, Bovolenta M, Richard I. Correction of exon 2, exon 2-9 and exons 8-9 duplications in DMD patient myogenic cells by a single CRISPR/Cas9 system. Sci Rep. 2024 Sep 11;14(1):21238. doi: 10.1038/s41598-024-70075-5. PMID: 39261505 Free PMC article.
- Poyatos-García J, Soblechero-Martín P, Liquori A, López-Martínez A, Maestre P, González-Romero E, Vázquez-Manrique RP, Muelas N, García-García G, Ohana J, Arechavala-Gomeza V, Vílchez JJ. Deletion of exons 45 to 55 in the DMD gene: from the therapeutic perspective to the in vitro model. Skelet Muscle. 2024 Oct 1;14(1):21. doi: 10.1186/s13395-024-00353-3. PMID: 39354597 Free PMC article.
- Heaton RA, Ball ST, Staunton CA, Mouly V, Jones SW, McArdle A, Jackson MJ. Peroxiredoxin 2 mediates redox-stimulated adaptations to oxidative phosphorylation induced by contractile activity in human skeletal muscle myotubes. Free Radic Biol Med. 2024 Dec 4;227:395-406. doi: 10.1016/j.freeradbiomed.2024.11.053. Online ahead of print. PMID: 39643135 Free article.
- Anwar S, Roshmi RR, Woo S, Haque US, Arthur Lee JJ, Duddy WJ, Bigot A, Maruyama R, Yokota T. Antisense oligonucleotide-mediated exon 27 skipping restores dysferlin function in dysferlinopathy patient-derived muscle cells. Mol Ther Nucleic Acids. 2024 Dec 21;36(1):102443. doi: 10.1016/j.omtn.2024.102443.